1.基本概念
http://d.hatena.ne.jp/teramonagi/ を参考にし、「R」を用いて、粒子フィルタ(詳しくは下記の参考文献を参考にされたい)の実行の仕方を説明する。
ここでは「Rcpp、inline」というパッケージを利用することにより、 「R」での計算を「C++」で実行させることによって粒子フィルタを実行させる。
2.インストール
1)Rtools.exeのインストール
http://cran.md.tsukuba.ac.jp/bin/windows/Rtools/ から「R」のバージョンにあったRtoolsをインストール。 (自分の場合は2.14.0)
2)「R」の再インストール
通常、「R」は「C:\Program Files\R\R-2.14.0」にインストールされていると思います。 しかしここではCのすぐ下である「C:\R\R-2.14.0」に再インストールする必要があります。
3)環境変数の設定 (システムのプロパティーから)
Pathのところに、次の3つを付け加える。
C:\Rtools\bin
C:\Rtools\MinGW64\bin
C:\R\R-2.14.0\bin
4)「Rcpp」と「inline」のテスト
Rを起動し、次のパッケージをインストール。
install.packages("Rcpp")
install.packages("inline")
library(Rcpp)
library(inline)
src <- '
Rcpp::NumericVector xa(a);
Rcpp::NumericVector xb(b);
int n_xa = xa.size(), n_xb = xb.size();
Rcpp::NumericVector xab(n_xa + n_xb - 1);
for (int i = 0; i < n_xa; i++){
for (int j = 0; j < n_xb; j++){
xab[i + j] += xa[i] * xb[j];
}
}
return xab;
'
fun <- cxxfunction(signature(a = "numeric", b = "numeric"), src, plugin = "Rcpp")
cygwin warning:
MS-DOS style path detected: C:/R/R-214~1.1/etc/i386/Makeconf
Preferred POSIX equivalent is: /cygdrive/c/R/R-214~1.1/etc/i386/Makeconf
CYGWIN environment variable option "nodosfilewarning" turns off this warning.
Consult the user's guide for more details about POSIX paths:
http://cygwin.com/cygwin-ug-net/using.html#using-pathnames
> fun(1:3, 1:4)
[1] 1 4 10 16 17 12
|
これは ”R上にRcppパッケージを使ったC++のコードを文字列として書いておいて、 それをinlineパッケージのcxxfunction関数を使用することによってそのままDLLとしてコンパイル、 更に自動でR上へロードしてそれをfun関数として割り当てる” ということを行っている。
5)再度環境変数の設定 (システムのプロパティーから)
4)で実行すると、エラーはでるものの結果は出ると思います。 その中のエラーをよく見ていると、 「CYGWIN environment variable option "nodosfilewarning" turns off this warning.」 と書かれており、 これに従い、再度環境設定すればよい。 よってCYGWINを新規作成し、nodosfilewarningとすればよい。 これで再度Rを起動させ同様のことをしても、エラーはなくなる。
3.サンプルテスト
次に、「RcppSMC」というパッケージをインストールする。
install.packages("RcppSMC")
ここでは最も簡単な線形の場合を 「simGaussian関数」を用いて、「線形ガウシアン状態空間モデル」を考察する。 よって次のモデルを考察していることとなる。
状態方程式: x(n)=x(n-1)+e(n)
観測方程式: y(n)=x(n)+f(n)
e,f はそれぞれ独立な標準正規分布に従う乱数。
> library(RcppSMC) 要求されたパッケージ Rcpp をロード中です 警告メッセージ: 1: パッケージ '‘RcppSMC’' はバージョン 2.14.2 の R の下で造られました 2: パッケージ '‘Rcpp’' はバージョン 2.14.2 の R の下で造られました > sim<-simGaussian(len=10) > sim $state [1] 0.5336825 1.4585169 3.8950859 2.7643721 2.4902512 3.5997874 3.2826262 [8] 4.3629772 3.7565205 4.1641225 $data [1] -0.00524223 1.45214378 2.76761464 4.12528078 2.55867236 3.83060418 [7] 2.05302909 4.85767138 3.93369179 4.17252643 |
stateがxでdataがyに対応し、 観測値であるyのみの値を使ってxを推定する場合に使われます。 このときには、blockpfGaussianOpt関数を使用します。 ただしこの関数は線形の場合のみ。
4.実行結果
> sim <- simGaussian(len=250) > res <- blockpfGaussianOpt(sim$data,lag=5,plot=TRUE) > lines(sim$state, lty=1, lwd = 3, col="red") |
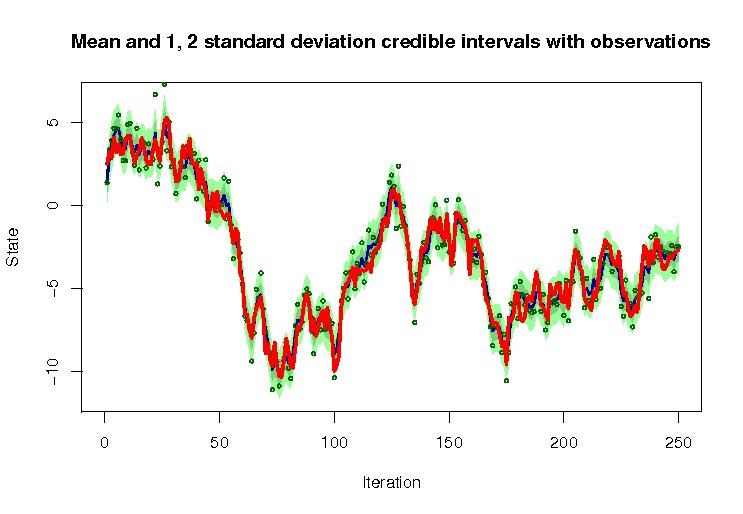
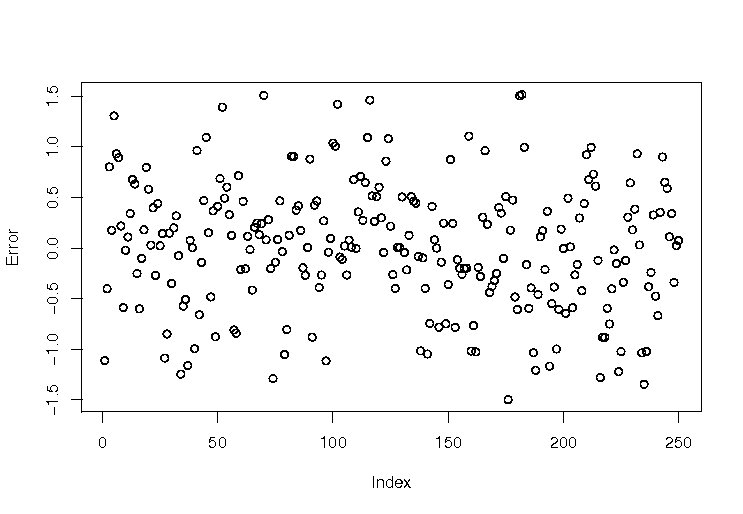
青が推定値、赤が実際の状態の値。 予測値の誤差は次のように計算することができます。
> error <- as.numeric(t(res$weight) %*% res$values / sum(res$weight)) - sim$state > plot(error, ylab ="Error") |
【参考文献】
時系列解析入門
予測にいかす統計モデリングの基本―ベイズ統計入門から応用まで
樋口知之(2011)データ同化入門
数理・計算の統計科学 (21世紀の統計科学) (第11章) 【FREE-PDF】
Sequential Monte Carlo Methods in Practice
Kitagawa, G. (1996), "Monte Carlo Filter and Smoother for Non-Gaussian Nonlinear State Space Models", Journal of Computational and Graphical Statistics, Vol. 5, 1-25.
北川源四郎 (1996)「モンテカルロ・フィルタおよび平滑化について」統計数理 Vol. 44. [PDF]
樋口知之 (1996)「遺伝的アルゴリズムとモンテカルロフィルタ」統計数理 Vol. 44. [PDF]
樋口知之 (2005) 「粒子フィルター入門」 [PDF]
C言語では、次を参照。
Particle Filter, Monte Carlo Filter
Back to R